TruSight Oncology UMI Reagents
TruSight Oncology UMI Reagents
Коррекция ошибок благодаря использованию уникальных молекулярных идентификаторов (UMI) для подготовки библиотек для секвенирования следующего поколения.
Циркулирующая опухолевая ДНК (ctDNA) может составлять очень маленькую фракцию свободной циркулирующей ДНК (cfDNA), уровни которой находятся около предела детекции метода NGS.
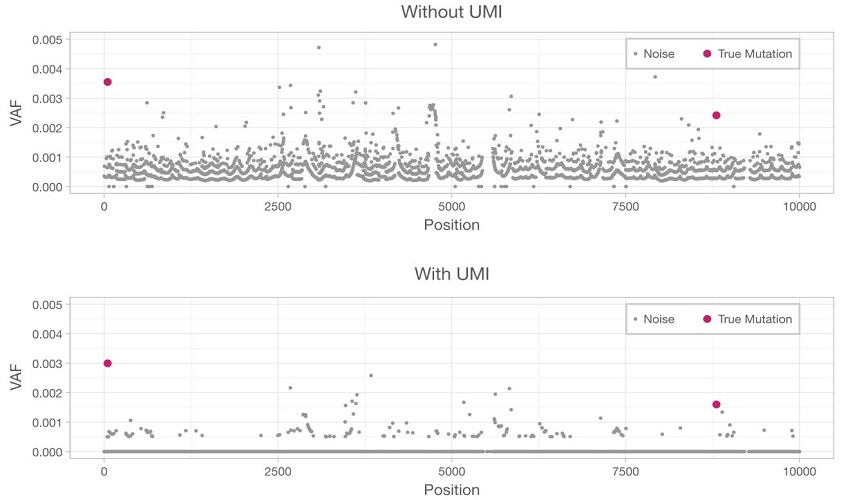
Набор реагентов TruSight Oncology UMI Reagents включает уникальные молекулярные идентификаторы (UMI), которые в тандеме с программным обеспечением UMI Error Correction App позволяют корректировать ошибки ПЦР и секвенирования и достоверно детектировать редкие мутации. Благодаря такому решению частота ошибок не превышает 0.007%.
Для протокола требуется 30 нг cfDNA в диапазоне 75–250 bp.
Ниже указаны католожные позиции, которые позволят приготовить cfDNA-библиотеки с использованием зондов TruSight Tumor 170 и отсеквенировать их на одной из высокопроизводительных систем Illumina:
-
TruSight cfDNA UMI for HiSeq 2500 (48 Samples) - реагенты для пробоподготовки и секвенирования 48 образцов на HiSeq 2500
-
TruSight cfDNA UMI for HiSeq 4000 (48 Samples) - реагенты для пробоподготовки и секвенирования 48 образцов на HiSeq 4000
-
TruSight cfDNA UMI for NovaSeq S2 (48 Samples) - реагенты для пробоподготовки и секвенирования 48 образцов на NovaSeq с ячейкой S2
-
TruSight Oncology UMI Reagents (16 indexes, 48 samples) - набор включает все реагенты для пробоподготовки кроме зондов для обогащения.
-
TruSight® Tumor 170 Content Set - зонды для обогащения (РНК* и ДНК)
* протокол TruSight Oncology UMI Reagents валидирован только для ДНК.
Спецификации
|
Время анализа |
1,5 дня |
|---|---|
|
Время ручного труда |
5 часов |
| Количество стартового материала | 30 нг cfDNA |
| Для работы с какими платформами подходит | HiSeq 2500, HiSeq 4000, NovaSeq 6000 |
| Тип нуклеиновой кислоты | ДНК |
| Специализированные типы образцов |
cfDNA |
| Метод | Targeted DNA Sequencing |
| Для исследования каких видов подходит |
Человек |
| Тип рака | Pan-Cancer |
| Технология | Секвенирование |
Снижение частоты ошибок с UMI
|
Количество запусков |
Средняя частота ошибок (Uncollapsed Reads) | Средняя частота ошибок (Collapsed Reads) |
|---|---|---|
|
1 |
0.038% | 0.0023% |
| 2 |
0.043% |
0.0024% |
| 3 |
0.035% |
0.0024% |
| 4 |
0.084% |
0.0019% |
Подготовка библиотеки проводилась с использованием реагентов TruSight Oncology UMI в сочетании с содержанием ДНК из панели TruSight Tumor 170 DNA, и 31 образец был распределен между четырьмя независимыми запусками на системе HiSeq 4000.
библиотек с помощью: